
The role of host proteins in SARS-CoV-2 replication
Abstract
L’infezione da SARS-CoV-2 altera l’omeostasi delle cellule ospiti, sfruttando diversi fattori dello stesso ospite, necessari per completare il ciclo di vita del virus. È stato scoperto che diverse proteine ospiti sono coinvolte nelle interazioni proteina-proteina con il complesso di trascrizione e replicazione virale (RTC) composto da tre proteine virali non strutturali (nsps): nsp12 (RNA polimerasi-RNA-dipendente), che media la polimerizzazione nucleotidica, e nsp8 e nsp7, agendo come primasi. In particolare, i risultati basati su studi proteomici e modelli computazionali hanno suggerito che le proteine leganti l’RNA come le proteine La-Related (LaRP) e le proteine leganti PolyA (PABP) potrebbero essere coinvolte nella replicazione del genoma virale.
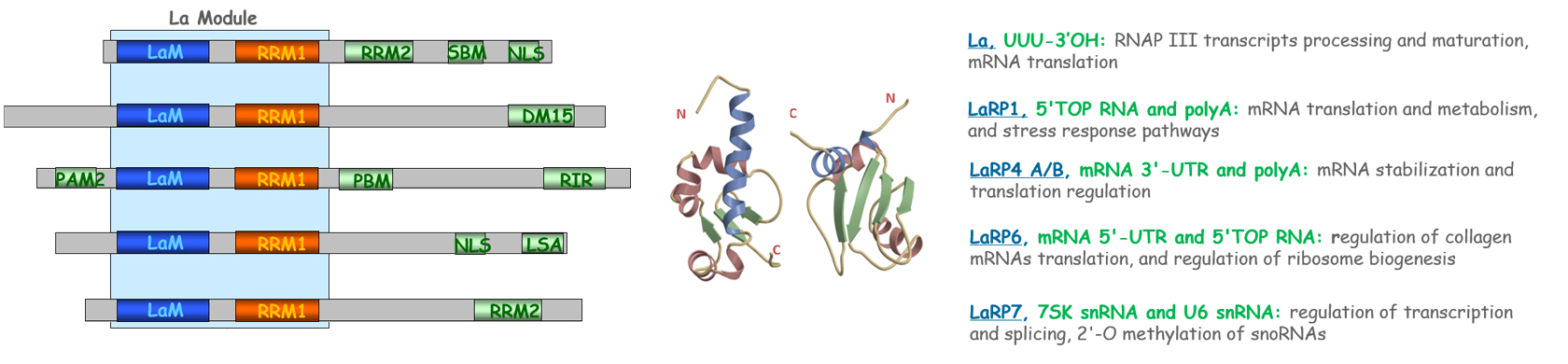
I membri della superfamiglia LaRP (Genuine La, LaRP1, LaRP4A, LaRP4B, LaRP6 e LaRP7), tutti caratterizzati da un motivo N-terminale estremamente conservato (La Module), riconoscono diversi substrati e sono coinvolti nella regolazione dell’ RNA cellulare. I PABP (PABPN1, PABPC1, PABPC3, PABPC4, PABPC5) sono anche coinvolti nella regolazione e nella traduzione dell’mRNA.
I nostri studi sono focalizzati sulla caratterizzazione biofisica e strutturale delle possibili interazioni che coinvolgono la triade nsp12-8-7 dell’RTC con fattori della cellula ospite utilizzando tecniche strutturali e biofisiche, come Bio-Layer Interferometry (BLI), Isothermal Titration Calorimetry (ITC), risonanza magnetica nucleare (NMR) e crio-microscopia elettronica (crio-EM). Abbiamo iniziato la nostra ricerca dalle LaRP per determinare se uno o più membri di questa peculiare superfamiglia sono cruciali per la replicazione virale. Fornendo informazioni cinetiche e strutturali, la nostra ricerca mira a contribuire allo sviluppo di nuovi approcci terapeutici.
Impatto
Nonostante nel maggio 2023 l’OMS abbia dichiarato la fine dell’emergenza sanitaria globale, il virus circola ancora consentendo l’emergere di nuove varianti e, potenzialmente, picchi di casi e decessi. Inoltre, l’ultima pandemia ha sottolineato l’importanza di approfondire la comprensione del meccanismo molecolare attraverso il quale i virus sequestrano e sfruttano i fattori dell’ospite. Questi studi sono, infatti, cruciali per sviluppare strategie efficaci contro gli agenti eziologici presenti e/o futuri, mirando alla prevenzione o mitigazione dei loro effetti sulla salute umana. In questo contesto, una conoscenza approfondita del ruolo svolto dalle proteine dell’ospite durante la replicazione virale contribuirà a comprendere le basi molecolari alla base dell’infezione da coronavirus.
Pipeline
-
CLINICAL
NEED -
DISEASES
ANALYSIS - DISCOVERY
-
PRECLINICAL
VALIDATION -
PRECLINICAL
DEVELOPMENT -
CLINICAL
STUDIES
Principal Investigator
Contatto
Aree terapeutiche:
Prodotto:
Farmaci chimici e biologici
Collaborazioni:
- Randall Division of Cell and Molecular Biophysics, King’s College London (KCL), Regno Unito
- Institute of Biochemistry I, University of Regensburg, Germania
Scarica il pdf del progetto
