Piattaforma di Ingegneria Tissutale
Descrizione
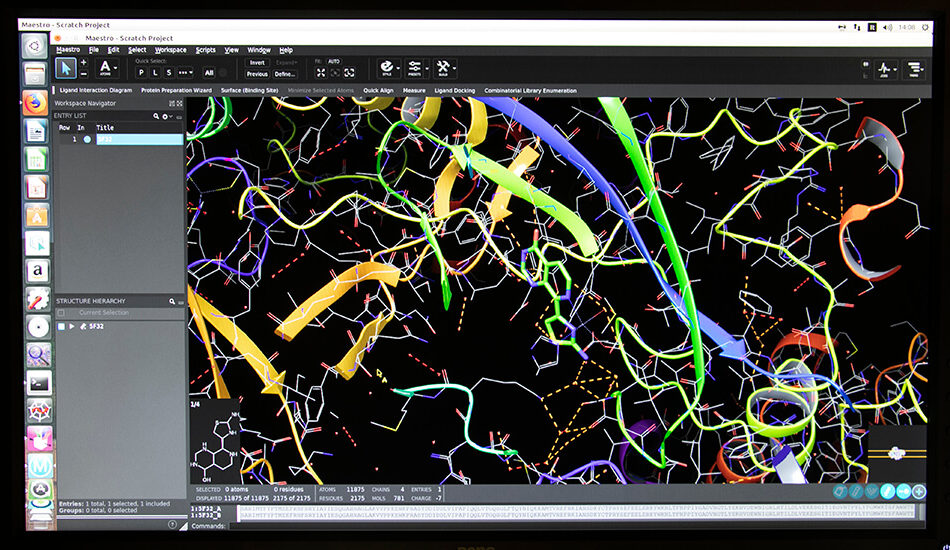
La piattaforma di ingegneria dei tessuti è impegnata su due fronti integrati della ricerca traslazionale: la ricerca di base e la ricerca applicata, in particolare in ambito cardiovascolare. Questa piattaforma consente la caratterizzazione meccanica e strutturale dei tessuti nativi e bioingegnerizzati e lo studio in vitro ed in vivo dello sviluppo di tessuto de novo. Inoltre, offre modelli strutturali deterministici per la crescita e la rigenerazione di tessuto e software customizzati per la caratterizzazione strutturale dei tessuti e per lo studio quantitativo di sezioni istologiche. Da un punto di vista traslazionale, la piattaforma di ingegneria dei tessuti si focalizza sulle applicazioni cliniche nel contesto delle malattie cardiovascolari, ponendo maggiore enfasi sullo sviluppo di medical device, sul loro posizionamento e sullo studio di meccanismi minimamente invasivi di deployment. In particolare, la piattaforma è focalizzata sullo sviluppo di cardiac patch, valvole cardiache ingegnerizzate, vascular graft e chordae tendineae bioingegnerizzate. I nuovi laboratori di ingegneria tissutale sono dotati di sistemi di ultima generazione e consta-no di due macro unità: la prima di biofabbricazione, la cui strumentazione include una piattaforma di bioprinting basata su un braccio robotizzato a sei gradi di libertà, strumenti per elettrofilatura ed elettrodeposizione, ed una di caratterizzazione dei materiali e costrutti ingegnerizzati che include macchinari per le prove meccaniche, per la caratterizzazione istologica e della micro e macro struttura
Competenze
- Sviluppo di dispositivi cardiovascolari come cardiac patch, vascular graft e valvole cardiache;
- Sintesi di polimeri con ampio grado di degradazione per lo sviluppo di dispositivi medici;
- Decellularizzazione di organi e tessuti;
- Caratterizzazione strutturale ed istologica di tessuti nativi ed ingegnerizzati;
- Sviluppo e prototipazione di sistemi per il posizionamento ed impianto in modalità minimamente invasiva dei dispositivi medici;
- Modellazione dei fenomeni che regolano la crescita e la rigenerazione del tessuto.
Dotazione tecnologica
Software
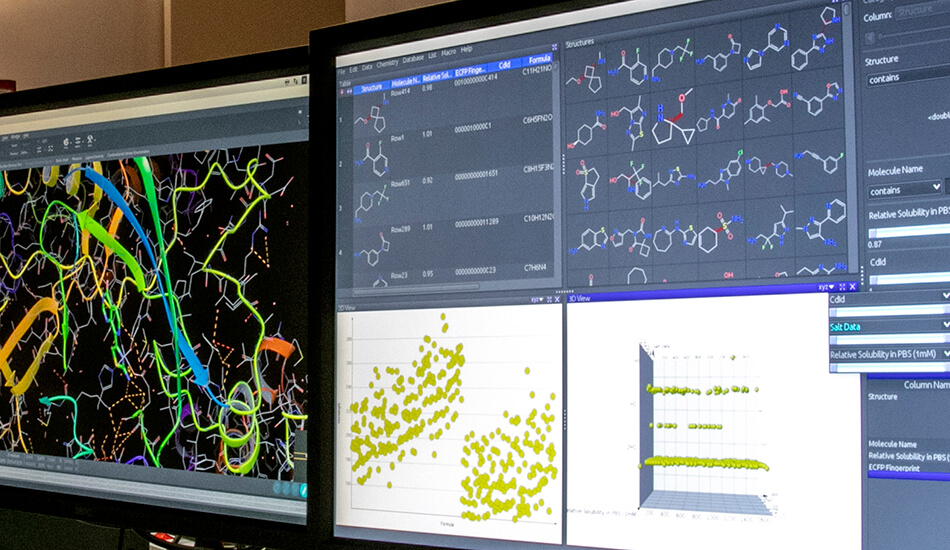
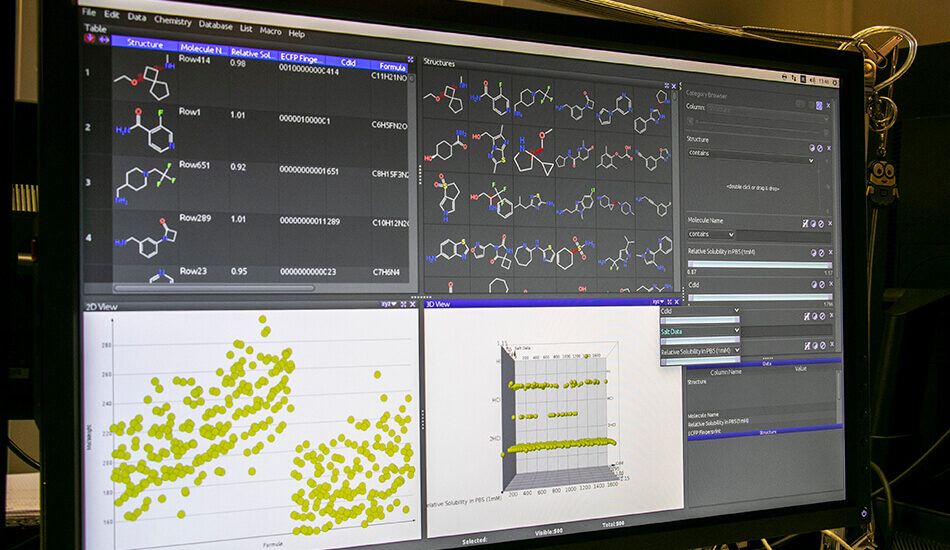
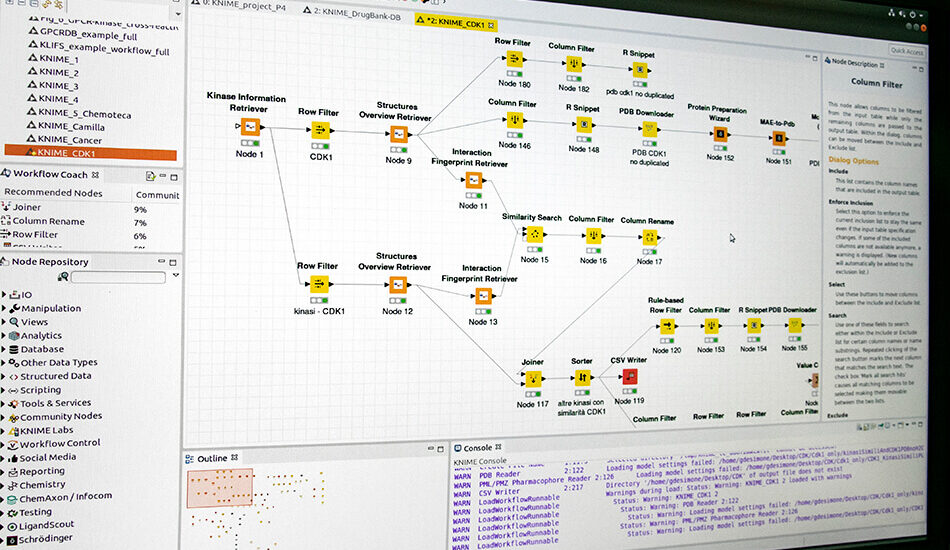
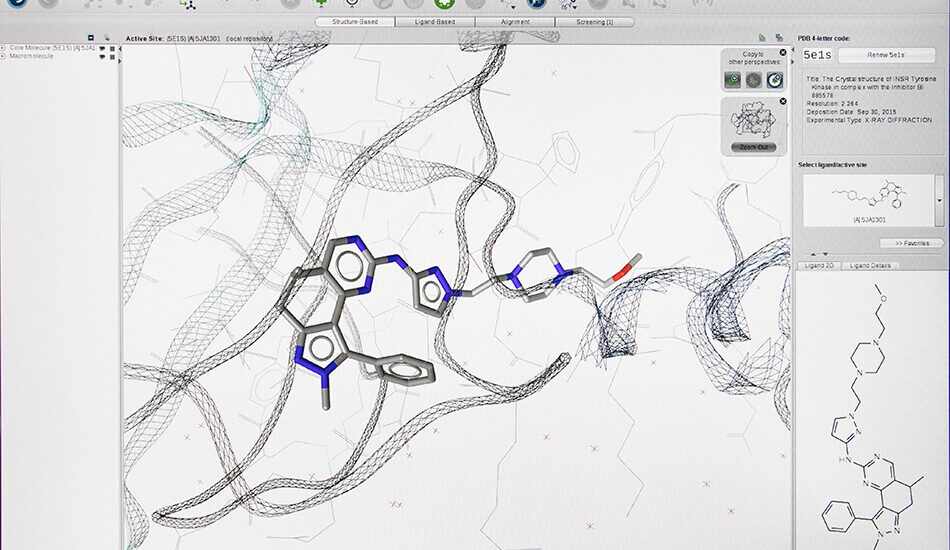
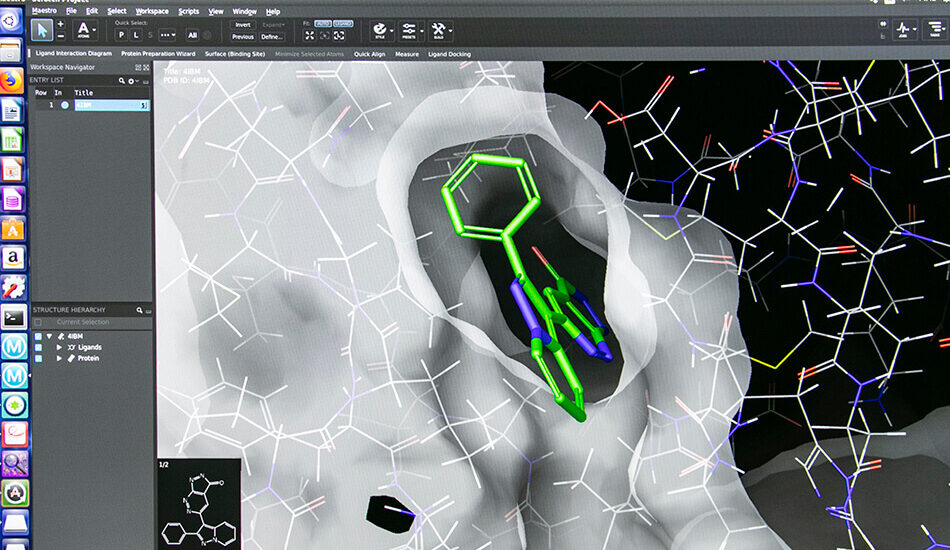
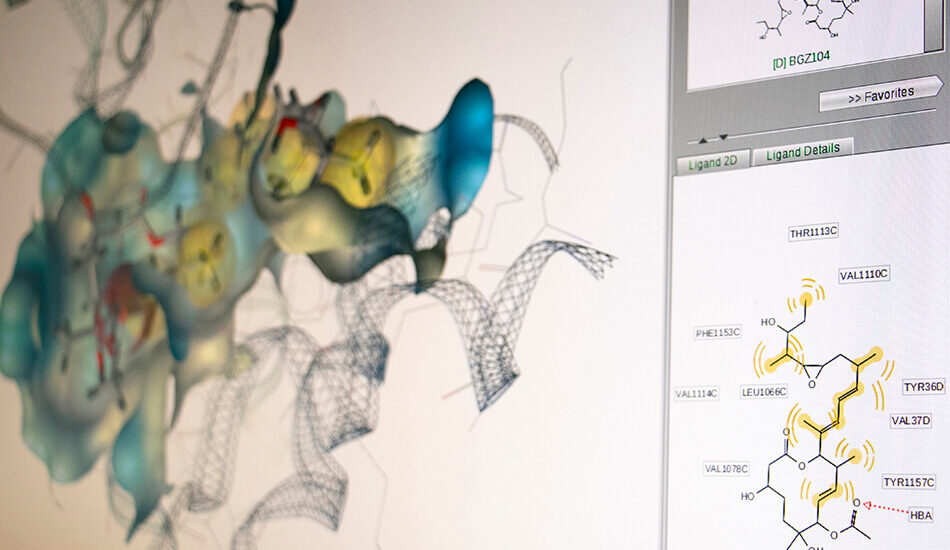
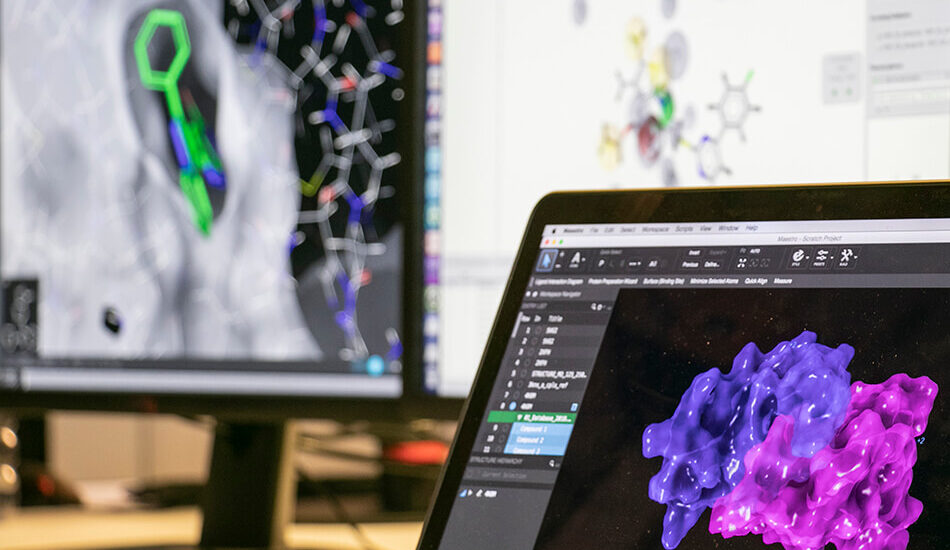
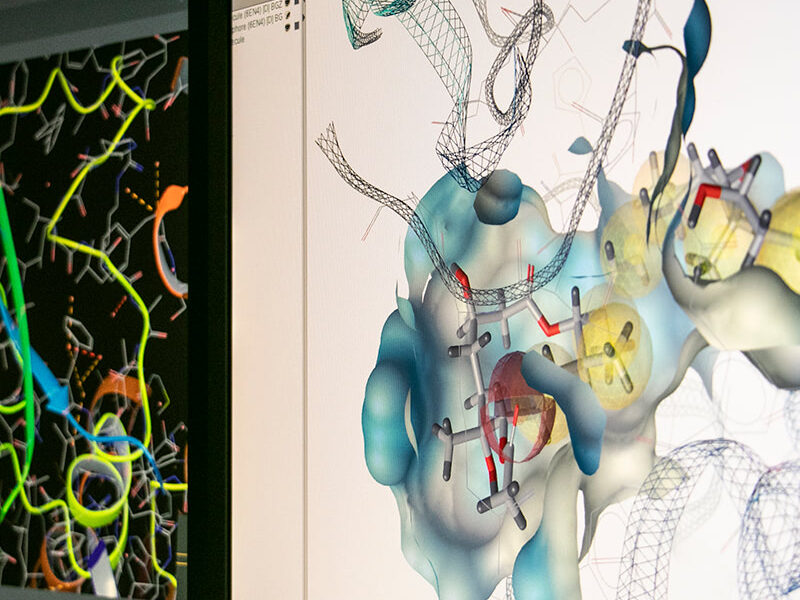
- Schrodinger suite for small molecule drug discovery;
- LigandScout expert suite;
- Autodock and Autodock Vina;
- Desmond (OPLS2005 and OPLS3e);
- AMBER;
- NAMD;
- VMD;
- Gromacs;
- RDKit;
- KNIME.
Hardware
- 6 Workstations;
- Server in HPC mode: 200 CPUs e 2 x NVIDIA Tesla K80.
Capacità di calcolo:
- Library optimisation → ~ 6,000 molecole/min
- Virtual Screening HTVS → ~ 5,000 molecole /min
- Virtual Screening SP → ~ 1,500 molecole /min
- Molecular Dynamics → ~ 200 ns/giorno/scheda (su un sistema medio di 40,000 atomi)
Piattaforma integrata in silico
Il gruppo sta realizzando una piattaforma integrata per lo studio dei network molecolari in collaborazione con il gruppo di bioinformatica.
Contatti:
Collaborazioni:
- University of Pittsburgh (PITT), Pittsburgh, USA
- University of Pittsburgh Medical Center (UPMC), Pittsburgh (PA), USA
- Politecnico di Milano, Milano, Italy
- Advances Solutions Life Sciences (ASLS) Inc. – Louisville (KY), USA
- IRCCS ISMETT, Palermo, Italy
- UAI, Argentina
- Aten, University of Palermo
- LivaNova – Mirandola, Italy
- University of Cincinnati, Cincinnati, USA
- West Virginia University, Morgantown, USA
- Harvard Medical School, Boston, USA
- University of Campinas, Campinas Brazil
- of Texas at Austin, Austin USA
- Telea Biotech – Tissue Engineering Biomedical Technologies, Italy
- University of Nagoya, Japan
- JOMDD, Tokyo, Japan
Hardware
I am text block. Click edit button to change this text. Lorem ipsum dolor sit amet, consectetur adipiscing elit. Ut elit tellus, luctus nec ullamcorper mattis, pulvinar dapibus leo.